用于DNA存储的纠错算法 | 文献学习

最近我在思考和选择合适的研究方向(也希望同时是合适的创业方向),觉得有多个选项都还不错,DNA存储应该算是其中一个。这是我很早时候起就比较感兴趣的,它愿景足够远大(把人类所有数据都保存下来,存放数百万年以上),技术足够创新(当然相应地,它离落地实用也还有一定距离,不用太担心还没学会就过时了),用到(或可能用到)的技术有我相对熟悉的(比如字符串处理及编解码等计算机算法),所替代的产品(当然是指计算机磁盘)我也并不陌生。总之,很适合 dive in!如所有其他科研工作者一样,要研究一个问题,就从读与之相关的文献开始。
今天学习的这篇,是去年九月农科院深圳农业基因组所阮珏课题组和潘玮华课题组发表在《国家科学评论(National Science Review)》杂志上的研究工作,题为 Improving error-correcting capability in DNA digital storage via soft-decision decoding。该研究开发了一个名为Derrick的软件(https://github.com/wushigang2/derrick),使用了一种软决策解码(soft decision decoding)算法,在不增加额外冗余度的条件下,用来提高DNA存储的纠错能力,从而增加潜在的存储容量,适用于 Illumina 、 PacBio 和 ONT 等测序平台。
DNA存储最终在应用上市时,其产品形态必然是软硬件结合的整体解决方案。但在当前阶段,该技术尚在研发早期,可以更侧重计算机编解码算法方面设计,并通过模拟计算的方式提前测试,“轻资产”地启动起来。加上大量的不同平台的公开原始测序数据可利用(用于深入探究不同测序平台的技术偏好和特点),在结合湿实验开展产品相关研发前,其实是有很多技术研发工作是可以提前进行起来的。这也是我个人看好这个技术方向的另一个重要原因。这篇文章,同领域内其他许多同类文章一样,也进行了这样的 in silico 的测试和评估。
DNA存储之所以被看好,是因为:一、其分子能保存巨大的信息量,相比传统磁盘等介质,其信息占用空间的效率(密度)简直是数量级的碾压;二、DNA的保存时长,从我们可以提取并研究数百万年前甚至更早的古生物的化石DNA,就可以直观了解;三、DNA的读取和写入,在今天可以方便地使用高通量测序仪和合成仪加以实现;四、DNA天然可以进行复制和其他分子实验操作。
然而,DNA的缺点也显而易见,在生物体内尚且经常发生突变,在分子实验和测序的过程中,每个环节也都会引入错误。即使错误率很低,但在天文数字的数据量基础上,这些错误就不容忽视了。通常的做法是以尽可能多的冗余方式来应对,因为错误和突变一般都是随机发生的,只要这些随机噪音被控制在一定范围,不把真实信号掩盖掉,DNA存储的信息就能通过合适的算法加以恢复。这就是这篇研究工作及其同类其他文章的逻辑基础。
这篇研究工作使用了信号处理工程中常用的RS纠错码和CRC64校验码。如文章图1a(见下图)所示,该算法首先使用多序列比对,把各个相似的分子叠合拼接到一起,获取一致序列(同时也评估每个碱基的可信度),这其实是典型的基因/基因组拼装过程,这个过程排除了绝大部分DNA分子或测序过程导致的错误。之后,每四列(或者说多序列比对对齐后的每四个碱基)作为一个RS块,进行校验和纠错,从而确保所存储信息的保真。与其他DNA存储算法所不同的地方是,本文在使用硬决策解码(hard decision decoding)失败后,会再次尝试使用软决策解码,这就使它相比其他已有算法,有更高的纠错能力。
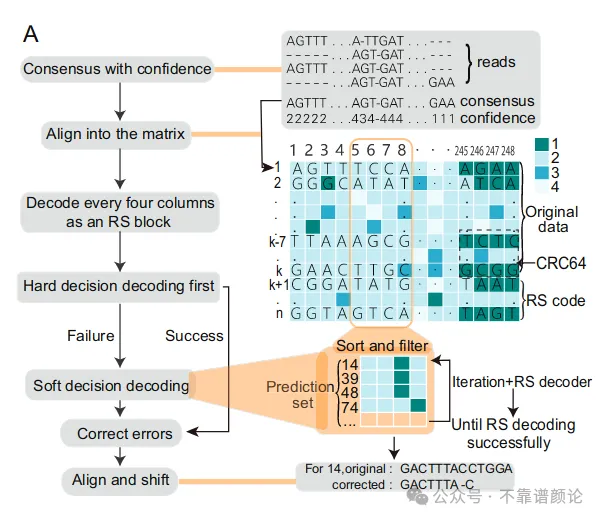
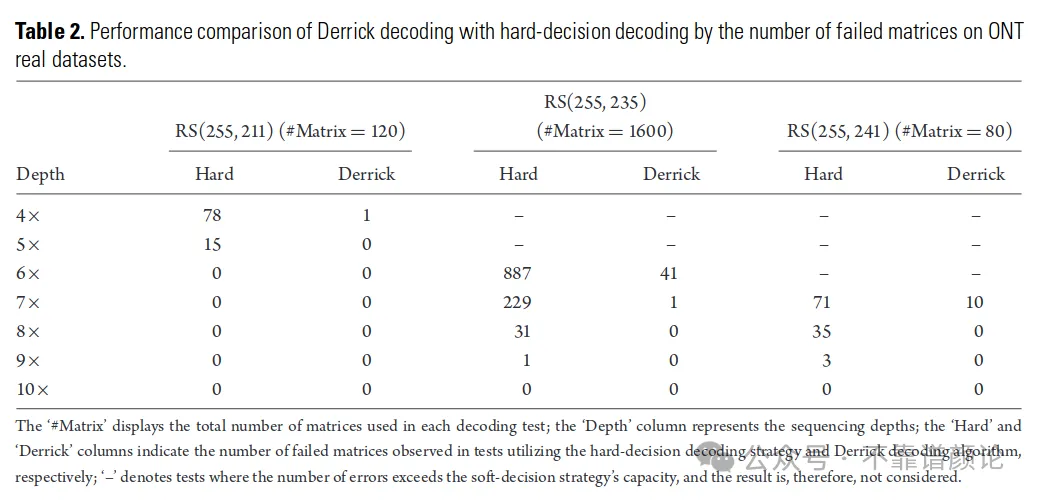
文章除了 in silico 的测试外,也通过人工合成核酸片段的方式,采用不同测序平台进行技术验证。下表展示了该软决策解码在ONT实测数据上带来的纠错能力的提升:
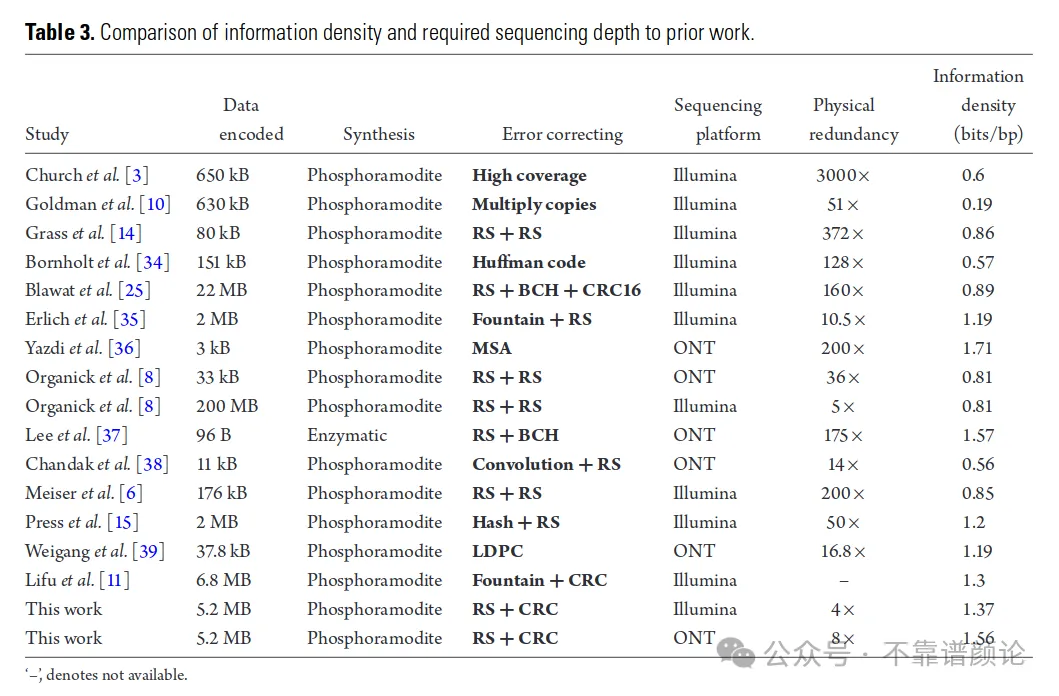
同时也和其他十多个不同算法进行了对比,展示了最高的信息存储密度:
这是一个很有趣的方向,在未来也会是一个非常实用甚至必需的方向。在高通量测序仪被大量应用于生命科学各个研究方向的今天,数据的产出速度仍在保持增长,这些数据是否能够一直被准确、高效、长期地存储下来,实际上是经常被我们忽略的问题。DNA存储技术的发展,有可能不仅是缓解,甚至很可能是彻底解决这个爆炸数据量的存储问题(我们只需要提供足够的核苷酸原料,外加相应的其他试剂即可)。到时候,生物数据只是从一种形态的DNA(待研究的、多样化的生物材料),转换成为另一种形态的DNA(统一的、模块化的生物存储),但能解决(至少部分解决)了珍贵生物材料难以长期保存的问题。
注:本文首发表于“不靠谱颜论”公众号,并同步至本站。